Décoder les mécanismes moléculaires qui contrôlent le développement et l'activation des cellules T
Les cellules T jouent un rôle crucial dans notre système immunitaire. Elles réagissent aux menaces extérieures en reconnaissant des parties spécifiques des agents pathogènes. Cette reconnaissance déclenche une chaîne de signaux intracellulaires, conduisant à différentes réponses immunitaires.
Compte tenu du rôle essentiel des cellules T, des efforts considérables ont été déployés au cours des dernières décennies pour cartographier et caractériser les mécanismes moléculaires responsables de la propagation des signaux à l'intérieur des cellules T.
Les signaux intracellulaires contrôlant l'activation des cellules T se déploient au travers de centaines de composants moléculaires soumis à de nombreuses régulations et pouvant interagir entre eux. Pour faire face à cette complexité, il est important de développer des stratégies d’analyses intégrées afin d'acquérir une compréhension globale des régulations moléculaires qui contrôlent les fonctions cellulaires et, par conséquent, les réponses immunitaires.
Romain Roncagalli et son équipe cherchent à comprendre les mécanismes moléculaires et les processus cellulaires qui déterminent le destin des cellules T dans diverses conditions physiologiques
Pendant des années, l'analyse des voies de signalisation des cellules T s’est vue limitée par les outils expérimentaux disponibles, et est restée restreinte aux protéines canoniques préalablement identifiées. De récentes avancées technologiques permettent maintenant de caractériser les réseaux protéiques et de déterminer l'abondance et les régulations post-traductionnelles de la majorité des protéines exprimées dans une cellule. Dans ce contexte, notre équipe développe des approches originales basées sur la spectrométrie de masse (MS) afin de générer des modèles aussi complets que possible et en y intégrant la dynamique des processus moléculaires associés aux états des cellules T.
En utilisant des modèles de souris génétiquement modifiées, nous étudions comment les protéines interagissent et sont modifiées en fonction des environnements dans lesquelles les cellules T évoluent. En particulier, nous cherchons à caractériser la stœchiométrie et la dynamique des interactions protéine-protéine (PPI) codant pour l'engagement du TCR et d'autres récepteurs exprimés à la surface des lymphocytes.
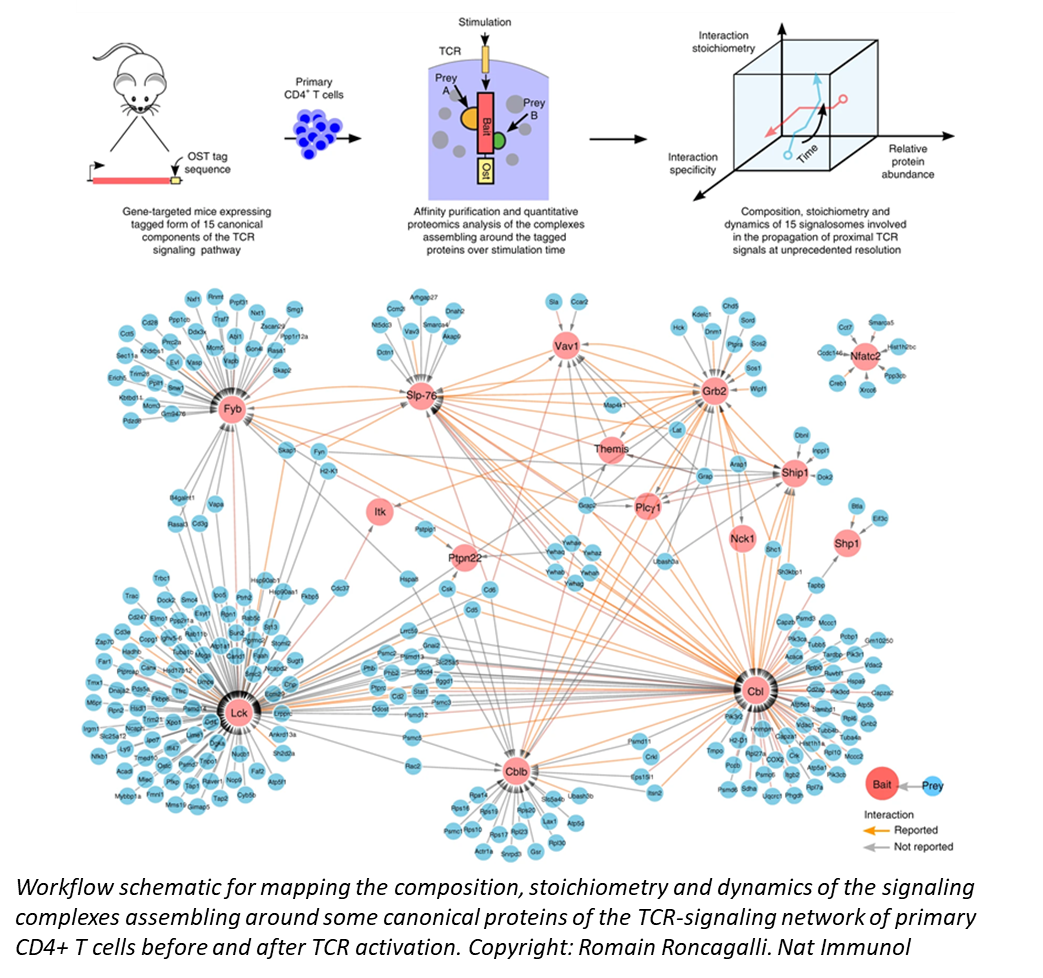
En utilisant des méthodes de phospho-protéomique, nous sommes également en mesure d'identifier, de quantifier et de caractériser la dynamique de phosphorylation de milliers de sites présents sur les protéines exprimées dans les cellules T primaires après la stimulation du TCR. Nos analyses révèlent une organisation cohérente des processus biologiques et la présence de modules fonctionnels associés au remodelage du cytosquelette, à la transcription, à la traduction et aux processus métaboliques, et ce durant les premières étapes de l'engagement du TCR. En utilisant des approches similaires, nous explorons également les mécanismes moléculaires qui permettent aux cellules T de différencier les antigènes en fonction de leurs affinités.

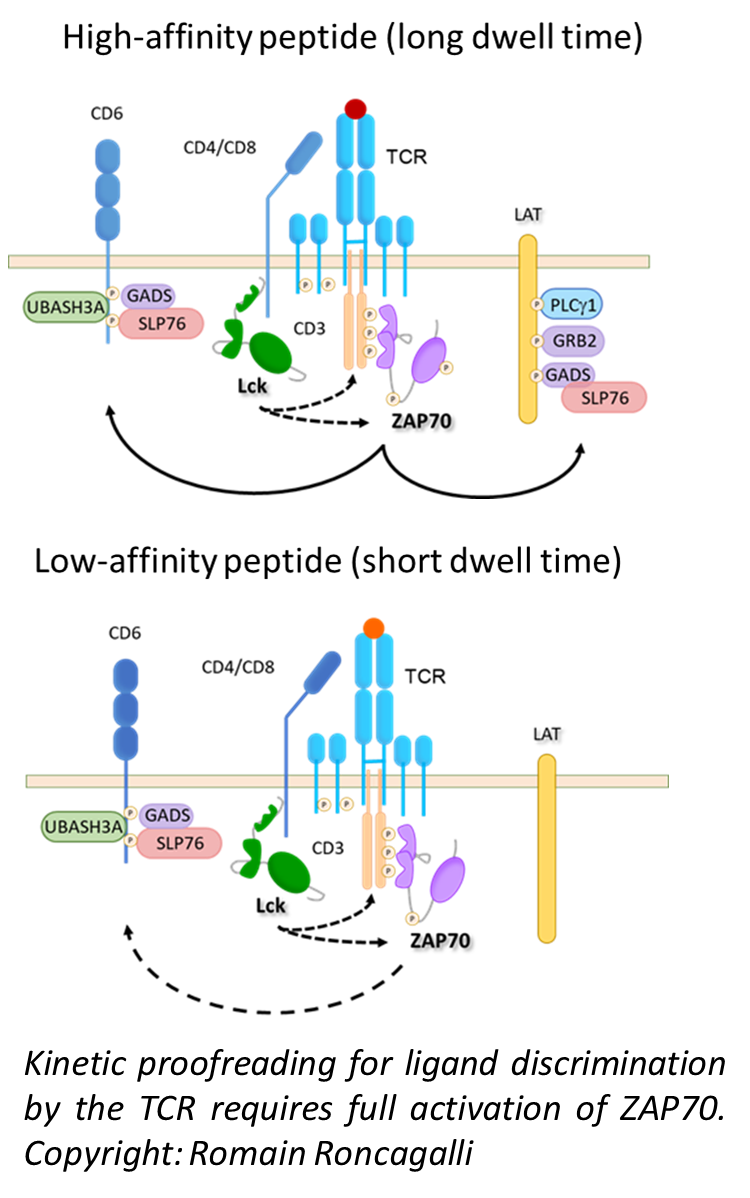
Au cours de nos recherches, nous nous sommes rendu compte que les mécanismes régulant la signalisation des cellules T, et donc leur destin, sont beaucoup plus complexes qu'on ne le pensait à l'origine, et que de nombreux composants moléculaires essentiels à la régulation des cellules T n'ont pas encore été caractérisés. Notre objectif est donc d'évaluer cette complexité à de multiples échelles, de la molécule à l'organisme vivant en passant par la cellule. Ainsi, notre stratégie de recherche consiste à combiner des méthodes dites « à haut débit » avec de l'analyse moléculaire ciblée, pour associer les régulations moléculaires aux phénotypes cellulaires qui émergent dans différentes conditions physiologiques.